Die Software zur Datenanalyse des GeoMx DSP ermöglicht eine einfache Visualisierung und Interpretation von Daten. Zusätzlich zu quantitativen Profiling-Daten können hochauflösende Bilder angezeigt werden. Dies ermöglicht eine Interaktion mit jedem Datensatz in Echtzeit.
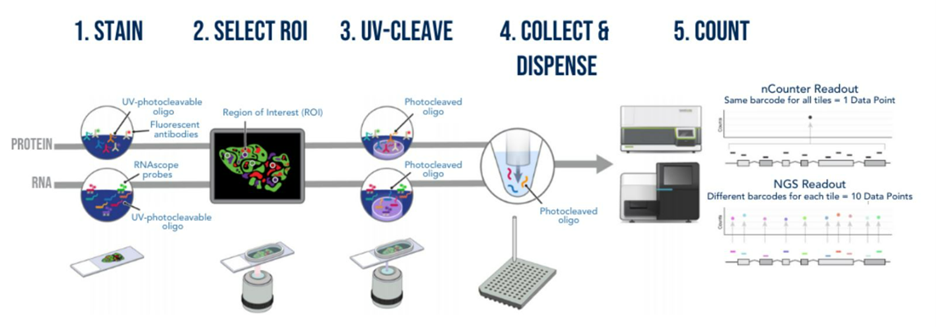
Wie funktioniert der GeoMx DSP?
Das Assay basiert auf RNA-Proben und spaltbaren Oligonukleotid-Tags. Im Anschluss an die Hybridisierung der Proben in formalinfixierte, in Paraffin eingebettete (FFPE) Gewebeschnitte werden die Oligonukleotid-Tags aus diskreten ausgewählten Bereichen des Gewebes mittels UV-Belichtung freigegeben. Die freigegebenen Tags werden anschließend mithilfe von RNAseq oder eines standardmäßigen nCounter-Assays quantifiziert und die Ergebnisse werden auf den Bereich des Gewebes angewandt. Dadurch ergibt sich ein räumlich aufgelöstes, digitales Profil der Menge an Analyten.1